No.08099 Re: glm()を用いたロジスティック曲線の当てはめ 【知ったかぶり】 2008/10/28(Tue) 09:44
result<-glm(cbind(dead,total-dead)~x,binomial)
ではないでしょうか?(参考書と一致しているとのことなので,ちょっと自信がありませんが)
>lines(x.jiku, exp(y.jiku), col="blue")
これは,family=poisson の場合.ロジスティック回帰の場合は,
lines(x.jiku,1/(1+exp(-y.jiku)),col="blue")
No.08100 Re: glm()を用いたロジスティック曲線の当てはめ 【青木繁伸】 2008/10/28(Tue) 11:13
No. 8099 の指摘の通りでしょう。( result <- glm(cbind(dead,total-dead)~x,binomial) )ま た,注意しないといけないのは,simplex 法によるあてはめは,F(x)=p[1]/(1+exp(p[2]+p[3]*x)) となっており,右辺の分子は1ではないこと,simplex 法では残差平方和を最小にする解を求めるようになっているのに対して glm は Deviance を最小にする解を求めるものであることです。生存率のように,0~1の値を取るもの(一般的には分布曲線)に曲線を当てはめるには glm を使うようにしましょう(simplex 法で F(x) の分子を1にしてもだめです)。
x.jiku <- seq(1.6, 1.9, 0.01)
y.jiku <- result$coefficients[1] + result$coefficients[2]*x.jiku
lines(x.jiku, 1/(1+exp(-y.jiku)), col="blue")
No.08101 Re: glm()を用いたロジスティック曲線の当てはめ 【波音】 2008/10/28(Tue) 11:59
知ったかぶり先生に青木先生,回答ありがとうございます。
提示した例において,当てはまりの良い曲線が描けなかったのは
lines(x.jiku,1/(1+exp(-y.jiku)),col="blue")
とすることによって解決できました。exp(y.jiku)ではなくて,1/(1+exp(-y.jiku))とするべきだったのですね。
> 生存率のように,0~1の値を取るもの(一般的には分布曲線)に曲線を当てはめるには glm を使うようにしましょう(simplex 法で F(x) の分子を1にしてもだめです)。
先 ほどglm()とsimplex()を使っていくつかの例を試していて,「見かけ上の当てはまりはよいけど,理論上ロジスティック曲線は限りなく1に近づ いていくはずだからsimplex()だとよくない場合があるのでは?」と質問しようとしたら,すでに回答が出ていました(^_^;)
> dat <- matrix(c(
+ 24, 1355,
+ 35, 603,
+ 21, 192,
+ 30, 224), ncol=2, byrow=TRUE)
> X <- c(0, 2, 4, 5)
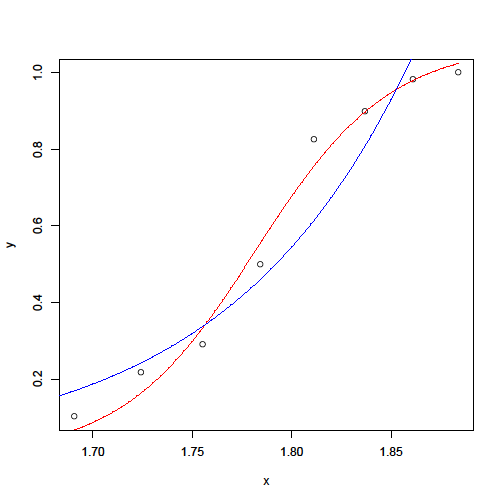
> plot(X, dat[,1] / dat[,2], xlim=c(0, 30), ylim=c(0, 1))
と, このようなデータセットの場合,見かけ上(特にx[0, 5], y[0, 0.2]の範囲でみると)当てはまりの良いのはsimplex()によって得られた曲線(添付図青いライン)ですが,この曲線だと「限りなく1に近づく」 ということはありえないということですね。
だから,これがもし致死量の算出の場合とかだったとすると,今回のデータセットをsimplex()で解析してしまうと正しいLD50が求められない,ということですね。
No.08102 Re: glm()を用いたロジスティック曲線の当てはめ 【波音】 2008/10/28(Tue) 12:05
ところで,なぜ青木先生がいつも添付する画像は(拡大画像でみても)あれほどキレイクッキリとした図になるのです か?pdf("sample.pdf")といったように,PDFデバイスを開いて描いても,それを.pngに変換すると本来のPDFほどの鮮明さが失われ てしまうのですが。。。
.pngの形式に変換しても,pdfの鮮明さを失わないようにするテクニックがあるのでしょうか?
No.08103 Re: glm()を用いたロジスティック曲線の当てはめ 【青木繁伸】 2008/10/28(Tue) 12:41
> なぜ青木先生がいつも添付する画像は(拡大画像でみても)あれほどキレイクッキリとした図になるのですか?
Macintosh ですが,添付する図は,R で表示されるグラフィックウインドウのスクリーンショットを取っています。PNG フォーマットの画像が得られます。クリックして表示されるのが原寸大の画像です。PNG も,原寸で表示するときが一番きれいです(PDF は拡大しても縮小してもいつもきれいですが。ちなみに,LaTeX に取り込むのは当然 PDF です)。特に何もしていないのですけれど。。それでもきれいだとすると,Macintoshのせいかな?
PDF を PNG にすると汚くなるのは,アンチエイリアシングとかアンシャープマスクとかの設定の差ではないでしょうか?線が細いとジャギーが目立ちますし。
No.08115 Re: glm()を用いたロジスティック曲線の当てはめ 【波音】 2008/10/29(Wed) 21:26
回答ありがとうございます。
今までPDFファイルを開いて,Adobe Readerの[ファイルをクリップボードにコピー]を経由してそれをコピーしていましたが,Windowsのプリントスクリーン機能でコピーしてやると,まったくラインがギザギザしたような感じにならなくなりました。
● 「統計学関連なんでもあり」の過去ログ--- 042 の目次へジャンプ
● 「統計学関連なんでもあり」の目次へジャンプ
● 直前のページへ戻る