R で処理するデータは,データフレームとして読み込まれる。データは,変数名,その変数の取る値,欠損値などさまざまな情報を持っている。これらの情報を全て定義しなくてもデータの分析を行うことはできるが,適切に定義しておくと,R での解析結果をそのまま報告書に含めることもできる。
たとえば,10人について,性別と血液型のデータがあるとしよう。 データフレーム(df) は,
df <- data.frame(sex = c(1,2,1,1,1,2,2,1,2,2), blood.type=c(1,2,3,4,1,2,3,3,2,1))のようにもできる。
> df sex blood.type 1 1 1 2 2 2 3 1 3 4 1 4 5 1 1 6 2 2 7 2 3 8 1 3 9 2 2 10 2 1しかし,このような単純な状態では,例えば性別の血液型の分布を求めようとしても,出てくる結果はわかりにくい。sex の1は男,2は女というのは常識的にわかるが,blood.type の1,2,3,4は何を表しているのかだろうか。そもそも,行と列の区別もわかりにくい。
> table(df$sex, df$blood.type)
1 2 3 4
1 2 0 2 1
2 1 3 1 0
データフレームを作るときに,
df <- data.frame(sex = c("男","女","男","男","男","女","女","男","女","女"),
blood.type=c("A","B","O","AB","A","B","O","O","B","A"))
のようにしておくと
> df
sex blood.type
1 男 A
2 女 B
3 男 O
4 男 AB
5 男 A
6 女 B
7 女 O
8 男 O
9 女 B
10 女 A
> table(df$sex, df$blood.type)
A AB B O
女 1 0 3 1
男 2 1 0 2
のように,結果がわかりやすくなる。しかし,男と女の順序とか血液型の順序が前の結果と違っていることにも気づく。sex blood.type 1 1 2 2 1 3 1 4 1 1 2 2 2 3 1 3 2 2 2 1のようにデータファイルが用意される。ついでに,変数の名前も後々のことを考えると日本語で付けておいた方がいいだろう(英語の論文・報告書を書く場合には別だが)。
性別 血液型 1 1 2 2 1 3 1 4 1 1 2 2 2 3 1 3 2 2 2 1このデータファイルを分析の対象とする。
2. R で分析をはじめる
まず,R を起動して,「その他」メニューで「作業ディレクトリの変更...」を選択し,ファイルの保存されているフォルダ ~/Desktop/example を選ぶ。
データを読むのは read.table 関数である。データファイルの一行目に変数名を書いたので,header=TRUE を指定する。
> df <- read.table("test.data", header=TRUE)
> df
性別 血液型
1 1 1
2 2 2
3 1 3
4 1 4
5 1 1
6 2 2
7 2 3
8 1 3
9 2 2
10 2 1
read.table("test.data", header=TRUE) のように,直接ファイルを指定して読み込む方法では,ファイルの文字コードが特定のものでないとエラーが起きることがある(Macintosh 場合は UTF-8)。そのような場合には,コネクションから読み込むようにする。
> con <- file("test.data", open="r", encoding="EUC-JP")
> df <- read.table(con, header=TRUE)
データのまとめをするために summary 関数を使ってみる。
> summary(df)
性別 血液型
Min. :1.0 Min. :1.00
1st Qu.:1.0 1st Qu.:1.25
Median :1.5 Median :2.00
Mean :1.5 Mean :2.20
3rd Qu.:2.0 3rd Qu.:3.00
Max. :2.0 Max. :4.00
ちゃんと日本語の変数名が書かれているが,性別の平均値とか血液型の平均値が取られていて変である。なぜそのようになるかと言えば,R は性別,血液型の取る値について何も知らされていないからである。> df[,1] <- factor(df[,1], levels=1:2, labels=c("男", "女"))
> df[,2] <- factor(df[,2], levels=1:4, labels=c("A", "B", "O", "AB"))
> df
性別 血液型
1 男 A
2 女 B
3 男 O
4 男 AB
5 男 A
6 女 B
7 女 O
8 男 O
9 女 B
10 女 A
factor 関数を使った2行で,データファイルを "男", "AB" のように入力したのと同じになった。そして summary 関数はさっきと異なる結果を返すようになる。
> summary(df)
性別 血液型
男:5 A :3
女:5 B :3
O :3
AB:1
単純な table 関数も,
> table(df[,1], df[,2])
A B O AB
男 2 0 2 1
女 1 3 1 0
のように,データの定義と同じ順序で結果が表示される。> tbl <- table(df[,1], df[,2])
> names(attr(tbl,"dimnames")) <- colnames(df)[c(2,1)]
> tbl
血液型
性別 A B O AB
男 2 0 2 1
女 1 3 1 0
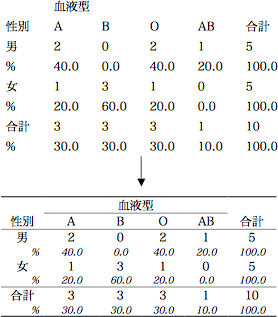
合計を作るには addmargin 関数が用意されている。
> tbl <- addmargins(tbl)
> tbl
血液型
性別 A B O AB Sum
男 2 0 2 1 5
女 1 3 1 0 5
Sum 3 3 3 1 10
Sum を 「合計」に変えるためには,列数と行数を求めて colnames, rownames 関数を用いる。
> nc <- ncol(tbl)
> nr <- nrow(tbl)
> colnames(tbl)[nc] <- rownames(tbl)[nr] <- "合計"
> tbl
血液型
性別 A B O AB 合計
男 2 0 2 1 5
女 1 3 1 0 5
合計 3 3 3 1 10
行方向の % を付けるのは,以下のようになる。> pcnt <- round(tbl/tbl[,nc]*100, 1)
> pcnt
血液型
性別 A B O AB 合計
男 40 0 40 20 100
女 20 60 20 0 100
合計 30 30 30 10 100
集計表と % の行を交互に書き出せばよい。
> cat("", colnames(df)[2], sep="\t"); cat("\n")
血液型
> cat(colnames(df)[1], colnames(tbl), sep="\t"); cat("\n")
性別 A B O AB 合計
> for (i in 1:nr) {
+ cat(rownames(tbl)[i], tbl[i,], sep="\t"); cat("\n")
+ cat("%", pcnt[i,], sep="\t"); cat("\n")
+ }
男 2 0 2 1 5
% 40 0 40 20 100
女 1 3 1 0 5
% 20 60 20 0 100
合計 3 3 3 1 10
% 30 30 30 10 100
命令と実行結果が入り交じってしまうが,以上の操作を関数で定義すれば,その問題は自然と解決される。
3. 関数の定義
関数の定義は以下のようになる。
shukei <- function(i, j, df)
{
tbl <- table(df[,i], df[,j])
tbl <- addmargins(tbl)
nc <- ncol(tbl)
nr <- nrow(tbl)
colnames(tbl)[nc] <- rownames(tbl)[nr] <- "合計"
pcnt <- round(tbl/tbl[,nc]*100, 1)
cat("", colnames(df)[2], sep="\t"); cat("\n")
cat(colnames(df)[1], colnames(tbl), sep="\t"); cat("\n")
for (i in 1:nr) {
cat(rownames(tbl)[i], tbl[i,], sep="\t"); cat("\n")
cat("%", pcnt[i,], sep="\t"); cat("\n")
}
}
shukei 関数は 3 つの引数を持つ。3 つめはデータフレームの名前,1つめは表側に来る変数のデータフレームでの列番号,2つめは表頭に来る変数のデータフレームでの列番号である。1列目と2列目の変数の集計は以下のようになる。
> shukei(1, 2, df) 血液型 性別 A B O AB 合計 男 2 0 2 1 5 % 40 0 40 20 100 女 1 3 1 0 5 % 20 60 20 0 100 合計 3 3 3 1 10 % 30 30 30 10 100大筋ができたので,shukei 関数の機能を拡張してみよう。まず,列方向にも % を計算できるようにして,引数で選べるようにする。また,% の数値が整数のときにも ".0" が付くようにする。
shukei2 <- function(i, j, df, row=TRUE)
{
tbl <- table(df[,i], df[,j])
tbl <- addmargins(tbl)
nc <- ncol(tbl)
nr <- nrow(tbl)
colnames(tbl)[nc] <- rownames(tbl)[nr] <- "合計"
if (row) {
pcnt <- tbl/tbl[,nc]*100
}
else {
pcnt <- tbl/rep(tbl[nr,], each=nr)*100
}
cat("", colnames(df)[2], sep="\t"); cat("\n")
cat(colnames(df)[1], colnames(tbl), sep="\t"); cat("\n")
for (i in 1:nr) {
cat(rownames(tbl)[i], tbl[i,], sep="\t"); cat("\n")
cat("%", sprintf("%.1f", pcnt[i,]), sep="\t"); cat("\n")
}
}
> shukei2(1, 2, df)
血液型
性別 A B O AB 合計
男 2 0 2 1 5
% 40.0 0.0 40.0 20.0 100.0
女 1 3 1 0 5
% 20.0 60.0 20.0 0.0 100.0
合計 3 3 3 1 10
% 30.0 30.0 30.0 10.0 100.0
> shukei2(1, 2, df, row=FALSE)
血液型
性別 A B O AB 合計
男 2 0 2 1 5
% 66.7 0.0 66.7 100.0 50.0
女 1 3 1 0 5
% 33.3 100.0 33.3 0.0 50.0
合計 3 3 3 1 10
% 100.0 100.0 100.0 100.0 100.0
shukei, sukei2 関数を定義しておけば,新たなデータを分析するときに必要な記述は,以下のような部分だけである。
df <- read.table("test.data", header=TRUE)
df[,1] <- factor(df[,1], levels=1:2, labels=c("男", "女"))
df[,2] <- factor(df[,2], levels=1:4, labels=c("A", "B", "O", "AB"))
:
shukei2(1, 2, df)
:
4. 出力結果をワープロに貼り込む
数個の表だけが必要ならば,コンソールに出力された結果をコピーペーストで Word などに取り込めばよい。分析表がたくさんできる場合は,
sink("出力ファイル名")
shukei(1, 3, df)
:
sink()
により,指定したファイルに結果が書き出されるので,まとめて Word に取り込めばよい。
5. LaTeX ソースを出力する
しかし,たくさんの表について,手作業で整形処理しているとうんざりしてくる。そこで,LaTeX のソースを出力するように関数を書き直す。書き直した関数のページを参照。
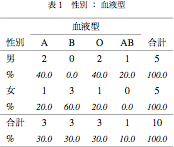
> cross(1,2,df)
\begin{table}[htbp]
\caption{性別 : 血液型}
\begin{center}
\begin{tabular}{lccccc} \hline
& \multicolumn{4}{c}{血液型}\\ \cline{2-5}
性別 & A & B & O & AB & 合計\\ \hline
男 & 2 & 0 & 2 & 1 & 5\\
\% & {\small \textit{40.0}} & {\small \textit{0.0}} & {\small \textit{40.0}} & {\small \textit{20.0}} & {\small \textit{100.0}}\\
女 & 1 & 3 & 1 & 0 & 5\\
\% & {\small \textit{20.0}} & {\small \textit{60.0}} & {\small \textit{20.0}} & {\small \textit{0.0}} & {\small \textit{100.0}}\\\hline
合計 & 3 & 3 & 3 & 1 & 10\\
\% & {\small \textit{30.0}} & {\small \textit{30.0}} & {\small \textit{30.0}} & {\small \textit{10.0}} & {\small \textit{100.0}}\\\hline
\end{tabular}
\\ \noindent
\end{center}
\end{table}
これを LaTeX でタイプセットする。