No.23020 Re: ピアソンの相関係数とコサイン類似度 【aoki】 2021/02/01(Mon) 15:15
コサイン類似度は
R で書くと
COS = function(a, b) sum(a * b) / sqrt(sum(a ^ 2)) / sqrt(sum(b ^ 2))ですね。
相関係数は,COS を使って書くと,
COR = function(a, b) COS(a-mean(a), b-mean(b))になります。
以下で検算
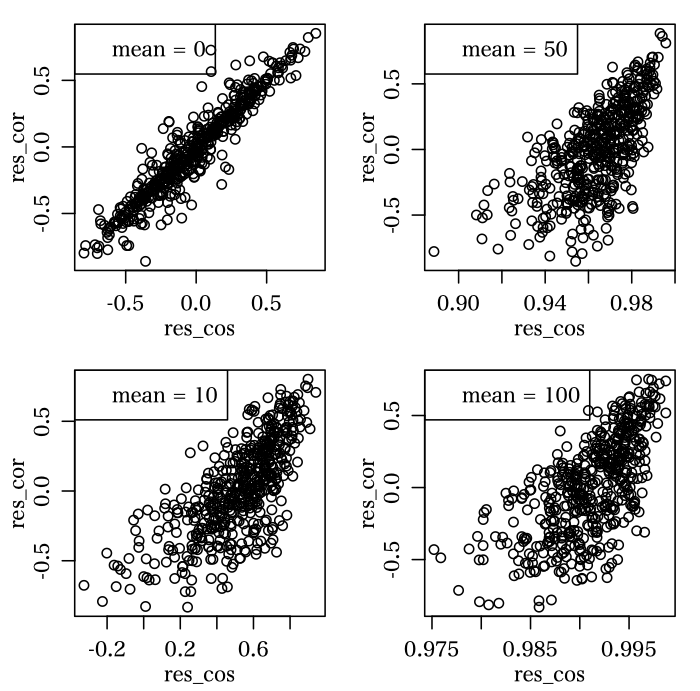
> a = c(1, 3, 2, 4, 5, 2, 3, 4, 5, 6)コサイン類似度は相関係数より大きめに出ていますね。
> b = c(2, 1, 2, 3, 5, 4, 3, 3, 3, 4)
> COS(a, b) # コサイン類似度
[1] 0.937391
> COR(a, b) # 相関係数
[1] 0.5477226
> cor(a, b) # 相関係数(いつも使っている関数)
[1] 0.5477226
大ざっぱに説明すると,コサイン類似度は原点からの距離,相関係数は平均値からの距離に基づくわけで,
> sum(a*b)のように,前者の方が大きいのです。
[1] 114
> sqrt(sum(a ^ 2))
[1] 12.04159
> sqrt(sum(b^2))
[1] 10.0995
> sum((a-mean(a))*(b-mean(b)))
[1] 9
> sqrt(sum((a-mean(a)) ^ 2))
[1] 4.743416
> sqrt(sum((b-mean(b)) ^ 2))
[1] 3.464102
それで,
> 114/12.04159/10.0995の違いになるのです。
[1] 0.9373918
> 9/4.743416/3.464102
[1] 0.5477226
この例だけだと,ふーんと思うだけかも知れませんが,
COS = function(a, b) sum(a * b) / sqrt(sum(a ^ 2)) / sqrt(sum(b ^ 2))のようなプログラムを書いてシミュレーションしてみるとちょっと驚くでしょう。
COR = function(a, b) COS(a-mean(a), b-mean(b))
sim = function(trial = 500, n = 10, mean = 50, sd = 10) {
res_cos = numeric(trial)
res_cor = numeric(trial)
for (i in 1:trial) {
a = rnorm(n, mean, sd)
b = rnorm(n, mean, sd)
res_cos[i] = COS(a, b)
res_cor[i] = cor(a, b)
}
plot(res_cos, res_cor)
legend("topleft", legend=sprintf("mean = %d", mean))
}
layout(matrix(1:4, 2))
par(mgp=c(1.8, 0.8, 0), mar=c(3, 3, 1, 1))
sim(mean=0, sd=10)
sim(mean=10, sd=10)
sim(mean=50, sd=10)
sim(mean=100, sd=10)
layout(1)
平均値が大きくなるにつれ(つまり,データのまとまりが原点から遠くにあるほど)コサイン類似度は大きくなります(当然ですが,相関係数はそんなことはありません)。
また,一般的には,コサイン類似度は対象間の類似度を表すために使われることが多いと思います。