No.15224 ROC解析について 【すずき】 2011/08/23(Tue) 11:33
統計初心者です。
他のホームページのスクリプトを用いてsplusで以下のようなスクリプトを入力して,このスク
リプトの実行を試みて,病気の有無と血中濃度のデータを用いてROC解析を行おうとしたのですが,なぜか病気の有無について0と1にならず,プロットが全
て0の方になっていて,すべて偽陽性の線上にのみプロットがあるという状態になってしまいます。(もちろん病気をもつ人もいて,病気ありを1,なしを0で
表しています。)
このような風になる理由がわからなくて,もしかしたらスクリプトが間違えっている可能性があるのではないかと思うのですが,もしどなたかわかる方いましたら教えてもらえませんか?
よろしくお願いします。menuROC <- function(data, disease, normal, nint=80)
{
disease <- data[[disease]]
# 疾患群
normal <- data[[normal]]
# ノーマル群
x <- c(disease, normal)
min.x <- min(x)
max.x <- max(x)
brks <- pretty(x, nint=nint)
# 集計区切り位置の計算
disease.f <- table(cut(disease, brks, left.include=TRUE))
# 疾患群データを集計
names(disease.f) <- NULL
disease.f <- c(disease.f, 0)
normal.f <- table(cut(normal, brks, left.include=TRUE))
# ノーマルデータを集計
names(normal.f) <- NULL
normal.f <- c(normal.f, 0)
result <- ROCfun(brks, disease.f, normal.f)
# 度数になったデータを使って,次の関数で計算
result
}
# 度数分布表から計算するための関数
ROCfun <- function(x, disease, normal)
{
distribution <- cbind(x, disease, normal)
k <- length(x)
n.disease <- sum(disease)
n.normal <- sum(normal)
Sensitivity <- c(1,(n.disease-cumsum(disease))/n.disease)
False.Positive.Rate <- c(1, (n.normal - cumsum(normal))/n.normal)
Specificity <- 1-False.Positive.Rate
plot(False.Positive.Rate, Sensitivity, type="b")
abline(h=c(0, 1), v=c(0, 1))
c.index <- 0 # area under ROC curve
for (i in 1:k) {
c.index <- c.index+(False.Positive.Rate[i]-False.Positive.Rate[i+1])*(Sensitivity[i+1]+Sensitivity[i])/2
}
result <- cbind(x, disease, normal, Sensitivity[-k-1], Specificity[-k-1], False.Positive.Rate[-k-1])
row.names(result) <- as.character(1:k)
names(result) <- c("Value", "Disease", "Normal", "Sensitivity", "Specificity", "F.P. rate")
result
}
guiCreate("Property",
Name = "ROCdata",
DialogPrompt = "Data",
DialogControl="List Box",
CopyFrom="TXPROP_DataFrames",
IsRequired=TRUE)
guiCreate("Property",
Name = "ROCcolumn1",
DialogPrompt = "Disease Data",
DialogControl = "List Box",
CopyFrom="TXPROP_DataFrameColumns",
UseQuotes = TRUE,
IsRequired=TRUE)
guiCreate("Property",
Name = "ROCcolumn2",
DialogPrompt = "Normal Data",
DialogControl = "List Box",
CopyFrom="TXPROP_DataFrameColumns",
UseQuotes = TRUE,
IsRequired=TRUE)
guiCreate("Property",
Name = "ROCnint",
DialogPrompt = "No. on intervals",
DialogControl = "String",
DefaultValue = 80)
guiCreate("Property",
Name = "ROCretval",
DialogPrompt = "Save As",
DialogControl="String")
guiCreate("FunctionInfo",
Name="menuROC",
Function="menuROC",
DialogHeader = "ROC",
PropertyList = "ROCretval, ROCdata, ROCcolumn1, ROCcolumn2, ROCnint",
ArgumentList = "#0 = ROCretval,#1 = ROCdata,#2 = ROCcolumn1,#3 = ROCcolumn2,#4 = ROCnint")
No.15225 Re: ROC解析について 【青木繁伸】 2011/08/23(Tue) 11:40
> 他のホームページのスクリプトを用いて
ちゃんと引用元を示して下さい(そのスクリプトを書いた人への礼儀でしょう)
また,追試を求めるなら,あなたが与えた情報より詳しいことが必要になるので,そのページを参照しないといけないでしょう。
No.15226 Re: ROC解析について 【青木繁伸】 2011/08/23(Tue) 11:44
R で動かすと,
エラー: 関数 "guiCreate" を見つけることができませんでした
となりますね。そのサイトでは guiCreate は定義されていますか?それとも,splus の標準関数なのでしょうか?
No.15227 Re: ROC解析について 【青木繁伸】 2011/08/23(Tue) 11:55
あなたは,データをどのように与えたのですか?
データの与え方はそのサイトに書いてませんでしたか?
プログラムを読んで,以下のようにしてみたらなんだか結果は出てきましたけど?
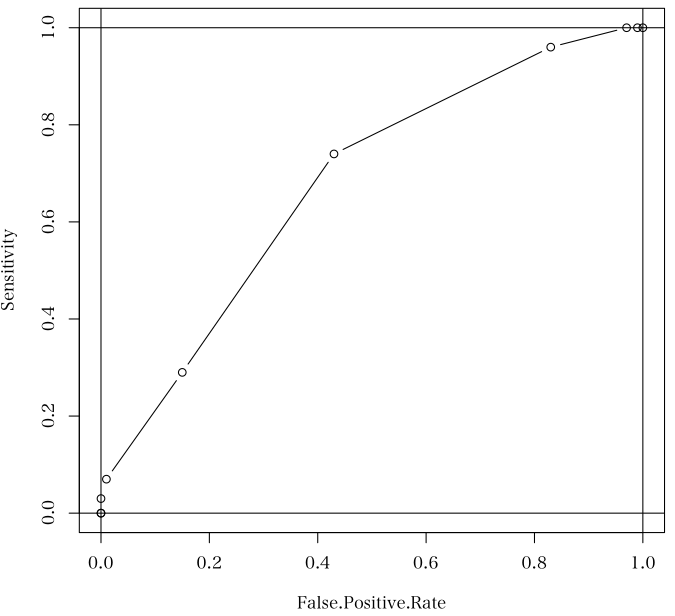
つまり,データはリストとして与え,分析対象を第2,第3引数で指定するんじゃないかな?> x <- list(disease=rnorm(100, mean=0.6), normal=rnorm(100))
> menuROC(x, "disease", "normal")
x disease normal
1 -4 0 1 1.00 0.00 1.00
2 -3 0 2 1.00 0.01 0.99
3 -2 4 14 1.00 0.03 0.97
4 -1 22 40 0.96 0.17 0.83
5 0 45 28 0.74 0.57 0.43
6 1 22 14 0.29 0.85 0.15
7 2 4 1 0.07 0.99 0.01
8 3 3 0 0.03 1.00 0.00
9 4 0 0 0.00 1.00 0.00
No.15228 Re: ROC解析について 【すずき】 2011/08/23(Tue) 12:44
青木先生,早急な回答ありがとうございました。
引用元について表記しておらず失礼いたしました。
上記のスクリプトは,
http://www.msi.co.jp/splus/support/salon/gui1.html
のものを引用させていただきました。
すみません,説明不足だったのですが,コマンドを実行するために,スクリプトを入力した後に,コマンドウィンドウでguiDisplayDialog("Function", "menuROC")と入力することによって,実行を行いました。
す
みません,ホームページを読んでもどのようにデータを与えたらいいのかよくわからなかったのでお聞きしたいのですが,今回作成されたROC解析のスクリプ
トでは,病気の有無を0と1で,血中濃度を連続変数で表し,これらの二つの変数の間でROC解析を行えないのでしょうか?
度々申し訳ないですが,回答よろしくお願いします。
No.15229 Re: ROC解析について 【青木繁伸】 2011/08/23(Tue) 12:50
> 病気の有無を0と1で,血中濃度を連続変数で表し,これらの二つの変数の間でROC解析を行えないのでしょうか?
できませんね。
どのようにデータを与えるかも,件のページで説明されています。
2. データの与え方
> データはデータ・フレーム(Excel ファイルをインポートしたものはデータ・フレームになります)を想定,そのうち1つの列が疾患群,別の1列がノーマルの生データとします
つまり,疾患群のデータ ノーマル群のデータ
123.456 324.769
: :
説明にはそうは書いてあるけど,普通は疾患群とノーマル群のデータ数は異なるので,データフレームでは無理。リストの方が一般的(データフレームはリストの一種)。
data <- list(疾患群=c(123.456, ...), ノーマル群=c(324.769, ...))
>
ここでのポイントは列の指定方法です。データ名は普通に指定するのに対して,2つの列の名前は文字列で指定します。つまり,メニューを使わずコマンド実行
させるなら,次のようになります。データ名を data,疾患群を疾患,normal群を非疾患という列の名前だったとして,
> menuROC(data, "疾患群", "非疾患群")
そいういうことで,やり直してみて下さい。
No.15239 Re: ROC解析について 【すずき】 2011/08/24(Wed) 10:42
青木先生,回答ありがとうございました。
おかげさまでROC曲線を作る事ができました。
もう一点だけ教えていただきたいのですが,このスクリプトでROC解析を行う場合,カットオフ値はどのように求めたらいいのでしょうか?
度々質問して申し訳ないですが,回答よろしくお願いします。
No.15243 Re: ROC解析について 【青木繁伸】 2011/08/24(Wed) 12:13
> カットオフ値はどのように求めたらいいのでしょうか?
ROC 曲線の解釈法そのままでは?つまり,左上隅に一番近い点。
No.15247 Re: ROC解析について 【すずき】 2011/08/24(Wed) 14:39
度々回答していただきありがとうございます。
説明不足ですみません。左上隅に一番近い点がカットオフ値を示すことはわかったのですが,その点からどうやって血中濃度のカットオフ値を求めることができるかがわかりませんでした。
度々質問して本当に申し訳ないのですが,回答よろしくお願いします。
No.15249 Re: ROC解析について 【青木繁伸】 2011/08/24(Wed) 16:14
> 左上隅に一番近い点がカットオフ値を示すことはわかったのですが,その点からどうやって血中濃度のカットオフ値を求めることができるかがわかりませんでした。
ROC 曲線は,カットオフ値を色々変えたときの特異度と感度をプロットしたものでしょう?どの点がどのカットオフ値かはわかるじゃないですか。
「統計数字を読み解くセンス」の187ページ(図10-2)参照。
No.15250 Re: ROC解析について 【青木繁伸】 2011/08/24(Wed) 20:14
R と splus の違いで,No. 15227 に示した結果のタイトルが表示されていなかったので,結果の読み方が分からなかったのかも知れません。
ROCfun の最後の方の二行を R に合うように変更します。 row.names(result) <- as.character(1:k)
names(result) <- c("Value", "Disease", "Normal", "Sensitivity", "Specificity", "F.P. rate")
以下のように変更
rownames(result) <- as.character(1:k)
colnames(result) <- c("Value", "Disease", "Normal", "Sensitivity", "Specificity", "F.P. rate")
ついでに menuROC の最初の方の一行も変更。
brks <- pretty(x, nint=nint)
を以下のように変更
brks <- pretty(x, n=nint)
そうして,以下を実行する> set.seed(1111)
> x <- list(disease=rnorm(1000, mean=1.5), normal=rnorm(1000))
> menuROC(x, "disease", "normal", nint=20)
Value Disease Normal Sensitivity Specificity F.P. rate
1 -3.0 0 5 1.000 0.000 1.000
2 -2.5 0 15 1.000 0.005 0.995
3 -2.0 0 45 1.000 0.020 0.980
4 -1.5 5 93 1.000 0.065 0.935
5 -1.0 17 150 0.995 0.158 0.842
6 -0.5 50 191 0.978 0.308 0.692
7 0.0 102 210 0.928 0.499 0.501
8 0.5 134 145 0.826 0.709 0.291
9 1.0 207 78 0.692 0.854 0.146
10 1.5 172 45 0.485 0.932 0.068
11 2.0 155 17 0.313 0.977 0.023
12 2.5 102 6 0.158 0.994 0.006
13 3.0 36 0 0.056 1.000 0.000
14 3.5 13 0 0.020 1.000 0.000
15 4.0 7 0 0.007 1.000 0.000
16 4.5 0 0 0.000 1.000 0.000
> abline(h=0.826, v=0.291, col=2, lty=3)
赤で示した行が図の水平垂直線が交わる点。カットポイントは 0.5
表
に示されている Value というのがカットポイントの候補。Disease 群は全部で 1000 例,Value が 0.5 未満のものは
(5,17,50,102) で計 174 例,0.5 以上のものは残りの 826 例。よって,Sensitivity は 826/1000 =
0.826 で赤で示した行の Sensitivity。Specificity は Normal 群の 0.5 未満が
(5,15,45,93,150,191,210) の 709 例を全例数の 1000 で割ったもの。709/1000 = 0.709
これが,赤で示した行の Specificity。1 から引いたものが False positive rate(F.P. rate)
0.291)。
図の赤線は,水平線は Sensitivity = 0.86,垂直線は F.P. rat e= 0.291。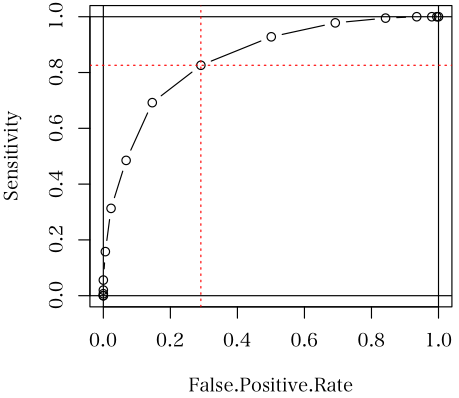
No.15270 Re: ROC解析について 【すずき】 2011/08/29(Mon) 10:03
返信が遅くなってすみません。
大変丁寧な回答をしていただきありがとうございました。
先生の著書のほうも読ませていただきました。
どうにかROC解析を行うことができるようになりました。
● 「統計学関連なんでもあり」の過去ログ--- 044 の目次へジャンプ
● 「統計学関連なんでもあり」の目次へジャンプ
● 直前のページへ戻る

